Wenn Viren Bakterien töten
In der sogenannten Phagentherapie werden Bakteriophagen eingesetzt; das sind Viren, die Bakterien infizieren und abtöten – eine vielversprechende Alternative zu Antibiotika, die wegen resistenter Bakterien an Wirksamkeit verlieren.
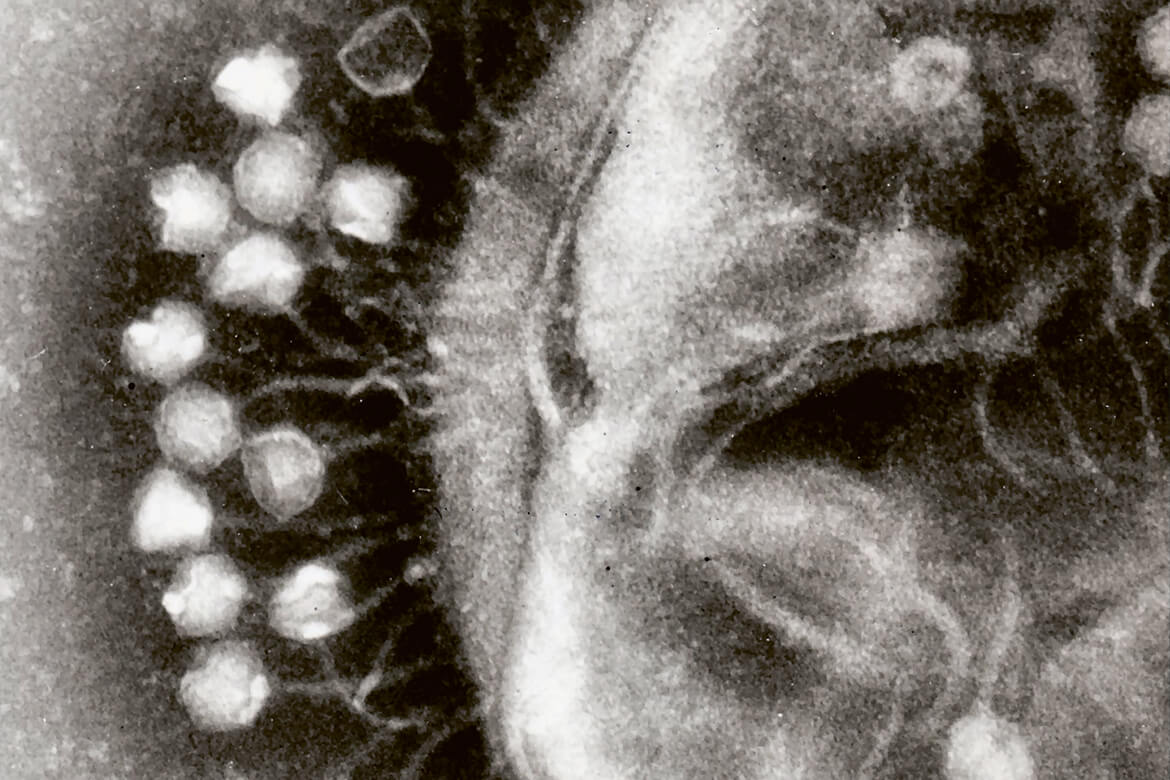
Hier docken Viren an die Zellwand eines Bakteriums an. Und können es ausschalten. | Bild: Wikimedia Commons/Dr Graham Beards
Phagen sind sehr wirtsspezifisch und müssen sorgfältig ausgewählt werden, damit sie ihr Ziel auch erreichen. Gegenwärtig erfolgt die Selektion mit langwierigen Tests im Labor. Um diesen Schritt zu beschleunigen, haben Forschende Computermodelle entwickelt, mit denen sich die Interaktionen zwischen Phagen und Bakterien auf der Grundlage ihrer Genome schnell und zu geringeren Kosten vorhersagen lassen. Die Studie wurde vom Schweizerischen Institut für Bioinformatik (ISB), von der Haute école d’ingénierie et de gestion des Kantons Waadt, der Universität Lausanne und dem Universitätsspital Bern gemeinsam durchgeführt.
Die Prognosemodelle beruhen auf der Methode des überwachten maschinellen Lernens – ein statistischer Ansatz, bei dem ein Algorithmus aufgrund von Daten lernt. «Wir haben Informationen zu mehr als tausend Interaktionen zwischen Phagen und Bakterien gesammelt, deren vollständige Genome bekannt sind», erklärt Carlos Peña vom ISB, der die Studie leitete. Die Forschenden suchten in diesen Genomen nach Merkmalen, die im Zusammenhang mit möglichen Wechselwirkungen stehen. Das kann zum Beispiel die Struktur bestimmter Proteine sein. Diese Daten wurden anschliessend verwendet, um den Algorithmus zu trainieren, der darauf Interaktionen mit einer Erfolgsquote von rund 90 Prozent korrekt prognostizieren konnte. «Die Vorhersagen müssen natürlich noch im Labor getestet werden. Da die Tests aber gezielter vorgenommen werden können, lässt sich viel Zeit sparen», resümiert Carlos Peña.




